Historia de la Lógica Transcursiva (Capítulo 108)
Cuaderno V (páginas 645 a 650)
(Dadas las referencias hechas, en varias oportunidades, al código genético tratando de hacer un símil con la estructura psíquica, es que hoy y durante algunos capítulos, nos acercaremos al aspecto biológico de la genética con algún detalle, con el fin de aclarar conceptos que, de otra manera, están careciendo de sustento. Trabajo tomado como referencia: "Química Bioquímica", Antonio Blanco, El Ateneo 2ª edición, 2000)
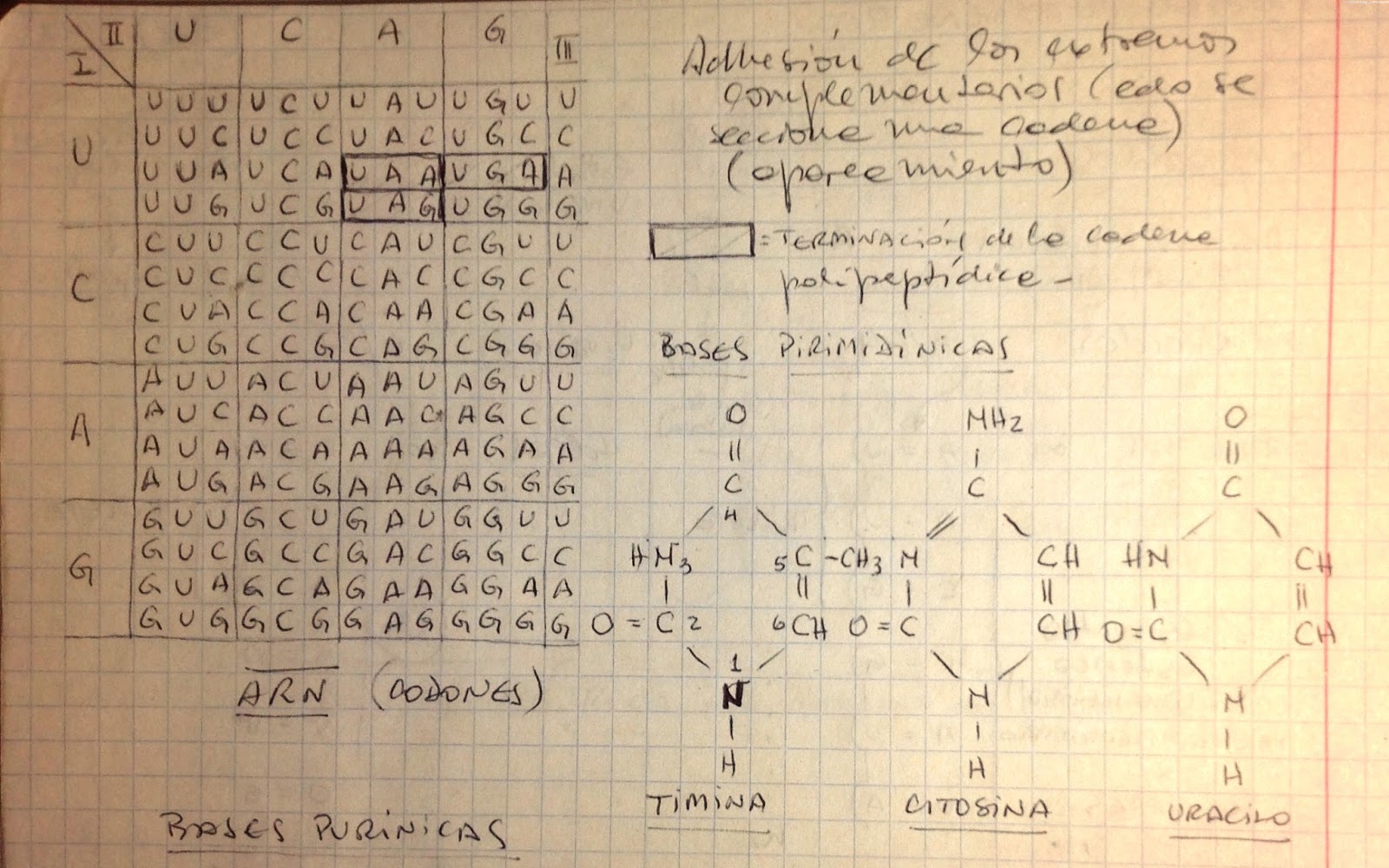
En las dos figuras anteriores podemos ver, en la superior a la izquierda, la tabla de los 'codones' (tripletes de bases) del ácido ribonucleico (ARN) con algún detalle operativo: el rectángulo que rodea a tres de los 64 codones está indicando que esas cadenas de tres bases no son un precursor de un aminoácido, sino una indicación de terminación de la cadena polipeptídica (luego ampliaremos sobre esto). A la derecha se puede ver el detalle químico de las bases pirimídicas (timina, citosina y uracilo), una de las dos tipos de bases nitrogenadas que componen en ADN.
En la figura inferior tenemos, en la parte de arriba, un detalle químico de las bases purínicas (adenina, guanina), las otras bases nitrogenadas que componen en ADN. En la parte de abajo muestra una tabla están encadenados los componentes que forman los nucleósidos (base nitrogenada + pentosa (azúcar)), y por otro lado, la formación de los nucleótidos (nucleósido + ácido fosfórico), que son los monómeros de los ácidos nucleicos (ADN y ARN), y son utilizados para proveer la gran energía que tienen acumulada en sus enlaces de fosfato. Las células poseen enzimas (proteínas especializadas) cuya función es, precisamente, romper estos enlaces y proveer la energía necesaria para el metabolismo celular.
En la figura anterior podemos apreciar una cadena de ADN (con un detalle de su química a la izquierda, donde se ven sus componentes: la base nitrogenada, el azúcar de cinco átomos de carbono, y el grupo fosfato). Las cadenas que componen el ADN (ilustración de la derecha) son complementarias y antiparalelas (una va en la dirección 5' → 3' y la otra 3' → 5')
La actividad del ADN es modulada por proteínas que se unen a él en lugares precisos. Su molécula no solo guarda información genética; también posee, como parte de su estructura, señales para el control de su propia actividad.
El ADN nuclear de las células eucariotas (con núcleo) está en los cromosomas, cada uno de los cuales, aloja una molécula de ADN (dos cuando se replica). Estas grandes moléculas están empaquetadas en complejos núcleo-proteínicos: la cromatina → profase → cromosomas (formaciones discretas con máxima densidad en la metafase).
El complejo núcleo-proteico tiene: ADN - Histonas - Polipéptidos con funciones estructurales - Proteínas regulatorias de la actividad genética y las Enzimas para la síntesis y procesamiento de los ácidos nucleicos. (figura)
En la cromatina, el ADN, a intervalos regulares da dos vueltas sobre el núcleo constituido por un octómero de histonas (las dos vueltas abarcan 146 pares de bases nitrogenadas)
Histonas + superhélice → Nucleosoma
Los Nucleosomas están conectados por un tramo de ADN de unas 50 bases de longitud. Estas estructuras se encuentran extendidas durante la internase; al iniciarse la mitosis (proceso de duplicación) se empaquetan en un 'solenoide' de 6 nucleosomas por vuelta.
El patrimonio genético o genoma de cada individuo está contenido en las moléculas de ADN de sus cromosomas y Mitocondrias (10%). En los cromosomas están las unidades de información: los genes, que son porciones de ADN que dirigen la síntesis de distintas especies de ARN funcionantes: Ribosomal (ARNr), de Transferencia (ARNt), Mensajero (ARNm) y otros.
Replicación ADN ⇾ transcripción ⇾ ARNm ⇾ traducción ⇾ Proteína
El ciclo celular (división) es regulado por un complejo de proteínas, ciclinas y quinasas dependientes de ciclinas, activadas por agentes externos (factores de crecimiento) que se unen a receptores específicos en la membrana plasmática y transmiten señales al interior de la célula. Fases: G1, S, G2, M. La replicación del ADN ocurre durante la fase S.
Replicación: en primer lugar se separan las dos cadenas, comenzando en un punto fijo: sitio de origen, en el cual, secuencias específicas sirven de señales de reconocimiento para las enzimas y factores que inician la replicación. En las eucariotas comienza la replicación en varios sitios. La separación se inicia simultáneamente en todos los cromosomas y en muchos puntos en cada uno de ellos. Así se producen, en la doble hélice, botones o 'burbujas' (replicones). En el humano se forman alrededor de 50.000 burbujas.
El código genético tiene validez universal, o sea, los codones (los tripletes de bases de la tabla) tienen el mismo significado en todos los seres vivos. Los genes que codifican para proteínas contienen información que, transcrita al ARNm, sirve para dirigir el ordenamiento de aminoácidos de los polipéptidos correspondientes. El conjunto de codones con la información completa para la síntesis de una cadena polipeptídica, se denomina: cistrón.
En los animales superiores hay un gran exceso de ADN, en relación a la necesidad de codificación, ya que el número de genes que dirige la producción de ARNm en el ser humano, es menor de 100.000. A diferencia de las bacterias, las células eucariotas no tienen la información genética dispuesta en trazos continuos de ADN, sino dividida en segmentos denominados: exones. Entre ellos están los intrones (sin información). Los intrones son más que los exones. (figura)
ADN Mitocondrial: las mitocondrias tienen su propio genoma, con un total de 16.569 pares de bases en el humano. Tiene 37 genes; 2 codifican el ARN ribosomal, 22 en ARN transferencia, y los otros 13, subunidades polipeptídicas componentes del complejo de la cadena respiratoria. Este genoma no es autosuficiente. La mayoría de las proteínas mitocondriales son codificadas en el núcleo y luego transportadas a la organela.
Diferencias con el ADN nuclear: 1) no posee intrones; 2) no procede en partes iguales de ambos padres, solo de la madre; 3) se divide independientemente del ADN nuclear; 4) su código difiere del universal (figura); y 5) tiene mayor tasa de mutaciones (10 a 100 veces más). No tiene buen sistema de reparación.
La figura muestra las diferencias entre los códigos del ADN nuclear y mitocondrial. Referencias: U = uracilo, G = guanina, A= adenina. Como vemos se dan distintas variantes: lo que en el nuclear es una terminación, en el mitocondrial es un aminoácido; lo que el nuclear es un aminoácido, en el mitocondrial es una terminación; finalmente, el mismo codón significa distinto aminoácido en un que en otro.
ARNm: Se sintetiza durante la transcripción y la secuencia de sus nucleótidos es complementaria con una del las 'hebras' del ADN guía (nuclear). Como el ARNm se sintetiza sobre una de las cadenas del ADN, se dice que la transcripción es 'asimétrica'. La hebra 'molde' de ADN sobre la cual se ensambla el ARN complementario es llamada: 'antisentido' o no codificante. La otra, no transcrita tiene la misma secuencia que el ARN sintetizado (admitiendo el cambio de timina (T) por uracilo (U)) y es la hebra 'con sentido' o codificante.
En las eucariotas se transcribe cada gen en toda su extensión. Este ARN es el ARNm 'precursor'. El proceso post-transcripción del ARNm no termina con el agregado del 'capuchón' de 7-metilGTP en el extremo 5' y de la cola de poliA (adenosina) en el 3'. (ver figura) La gran mayoría del ARNm primario contiene secuencias intercaladas o intrones, que es necesario eliminar. Este proceso se llama 'splicing'. Se seccionan los intrones y luego se empalma en el orden correcto, los exornes para formar el ARNm 'maduro'. Los intrones tienen una extensión de 60 a 10.000 pares. Todos empiezan con las bases GU {1000 en binario, según la siguiente convención: C = 01, G = 10, A = 11, T = U = 00} (extremo 5'), y terminan en AG {1110} (extremo 3'). Hay otra secuencia 'condenso' situadas entre -20 y -50 del extremo 3' (aguas arriba) del intrón llamado 'ramificación' (UACUAAC en las levaduras).
Una vez formado el ARN maduro o mensajero, es exportado al citoplasma a través de los poros de la membrana nuclear.
La molécula de ARNm está formada por el 'capuchon' de 7-metilGTP en el extremo 5', ⨁ una secuencia no codifican (de más de 100 bases) hasta llegar al codón de iniciación, que siempre es AUG (metionina) ⨁ la porción codificante (sucesión de codones que indica la secuencia de aminoácidos en la proteína a sintetizar) ⨁ codón de terminación (UAA, UAG o UGA) ⨁ segmento de longitud variable ('trailer') ⨁ una 'cola' PoliA de 200 unidades de Adenina (en la mayoría de los ARNm). Las últimas dos porciones no son traducibles. El trozo codifican se denomina: 'encuadre' o 'marco de lectura abierto.
ARNr (ribosomal): integra los ribosomas. Las células de los mamíferos tienen una 1010 ribosomas; las bacterias 20.000. El ARNr maduro se une a las proteínas para constituir las subunidades de los ribosomas. Cuando las dos porciones se unen para formar la partícula ribosomal, queda entre ellas una hendidura por donde se desliza ('lector') el ARNm durante la síntesis de proteínas. En la partícula completa se reconocen dos sitios adyacentes. A (Aminoacil) y P (Peptidil) a las que se unen las moléculas de ARNt cargadas con aminoácido.
ARNt (transferencia): llamado soluble es el encargado de unirse a aminoácidos (aa) libres en el citosol y transportarlos hasta el lugar de us ensamble. Existen ARNt específicos para cada aa. Su extremo 3' se une al aa y es idéntico en todos los ARNt; la secuencia terminal es siempre CCA. La especificidad del ARNt radica en su anticodón; triplete complementario del codón del aa transportado. El apareamiento codón-anticodón [la LT propone una tabla y un método para encontrar el anticodón de cualquier codón a traves de seis ejes de simetría] es antiparalelo y la secuencia se lee en el sentido 5'→ 3'. Así GUA (anticodón)↔︎UAC (codón Tirosina), GGC (anticodón)↔︎GCC (codón Alanina).
El ARNt actua como una molécula intermediaria o adaptadora que reconoce una secuencia determinada de nucleótidos en el ARNm (codón) y permite ubicar en su sitio el aa correspondiente. ☛El ARN puede almacenar información genética y además catolizar su propia replicación.☚
Biosíntesis de las proteínas (traducción):
① Activación de aminoácidos: requiere aa libres, ARNt sintetasas, ARNt, ATP y Mg++.
El aa reacciona co el ATP → aminoacilAMP-enzima. El aa activado es transferido al ARNt específico.
② Iniciación: varios factores de iniciación interaccionan con el capuchón del extremo 5' del ARNm. También se unen subunidades ribosomales menores y otros, y forman el 'complejo de preiniciación'. Este complejo se desplaza sobre el ARNm en sentido 5' → 3' hasta ubicar el codón de iniciación AUG (Metionina), el cual se aparea al anticodón (CAU) del ARNti, junto a otros agregados, se adhiere al sitio P. Se forma el complejo de iniciación.
③ Elongación: es un ciclo que se repite con cada aa agregado. Se une al sitio A del ARNt, cuyo anticodón se acople al codón del ARNm → formación unión peptídica y translocación: el grupo carboxilo de la Metionina del ARNti; en el sitio P se une a la función amina del aa del ARNt en A. Se forma un dipeptidil que queda unido al ARNt ubicado en A. El ARNti del sitio P descargado de su Metionina es liberado. El dipeptidil se desplaza de A → P. El ribosoma avanza al siguiente codón sobre el ARNm en el sentido 5' → 3' y se repite el ciclo. Pasados varios ciclos, la cadena peptídica unida al ARNt en el sitio P ha crecido. La proteína se ensambla desde el extremo N al C-teminal. La cadena es transferida al ARNt en A.
④ Terminación: una vez completada la cadena se llaga a un codón de terminación (UAA, UAG o UGA) desconocido por el factor de liberación asociado a GTP. La cadena polipeptídica es separada del ARNt por hidrólisis. Se liberan el ARNt descargado y las subunidades ribosómicas. Una cadena de ARNm dirige simultáneamente la síntesis de varias moléculas de proteína. El conjunto de ocho o más ribosomas sobre el ARNm se llama 'polisoma'. La cadena debe plegarse sobre sí misma para adquirir una estructura adecuada (funcional). Este plegamiento es asistido por las 'chaperonas'.
Mutaciones genéticas: mutación es cualquier cambio en la secuencia del ADN.
a) Mutaciones puntuales: reemplazo de una base por otra, o la adición o pérdida de un par de bases. Sustitución: se cambia una base por otra del mismo tipo → Transcripción; se cambia una base por otra de otro tipo → Transversión. La sustitución de bases o la pérdida o adición puede perderse el marco de lectura abierta.
b) Cambios estructurales en los cromosomas: alteran una porción más o menos amplia del cromosoma y alteran el orden lineal del ADN.
1) Deleción: pérdida de un segmento de ADN.
2) Duplicación: repetición de un trozo de ADN.
3) Inversión: cambio de sentido de un tramo dentro de la molécula de ADN.
4) Traslocación: un segmento de una molécula de ADN cambia de posición relativa dentro del mismo cromosoma o se incorpora a otro.
Mutaciones somáticas y germinales: las mutaciones ocurren al azar en cualquier sitio del genoma y en cualquier célula del organismo. Las somáticas afectan al tejido que forman. Si la reproducción se hace por cruzamiento sexual la mutación desaparece con la muerte del organismo. Las mutaciones de las células germinales se transmiten a las nuevas generaciones; estas constituyen en última instancia, el origen de la variabilidad genética existente [esto es un prejuicio del neodarwinismo] en las poblaciones. Las mutaciones dan origen a formas alternativas del mismo gen, denominados 'a lelos'. En los organismos diploides {con doble número de cromosomas, a diferencia de los gametos que tienen una sola serie} los descendientes reciben, para los genes que ocupan el mismo locus {lugar} en un par de cromosomas homólogos, un alelo materno y otro paterno. Si uno es normal y el otro mutante: 'heterocigota' para ese caracter, si ambos son normales o ambos tienen igual mutación: 'homocigota'.
¡Nos vemos mañana!
(Dadas las referencias hechas, en varias oportunidades, al código genético tratando de hacer un símil con la estructura psíquica, es que hoy y durante algunos capítulos, nos acercaremos al aspecto biológico de la genética con algún detalle, con el fin de aclarar conceptos que, de otra manera, están careciendo de sustento. Trabajo tomado como referencia: "Química Bioquímica", Antonio Blanco, El Ateneo 2ª edición, 2000)
En las dos figuras anteriores podemos ver, en la superior a la izquierda, la tabla de los 'codones' (tripletes de bases) del ácido ribonucleico (ARN) con algún detalle operativo: el rectángulo que rodea a tres de los 64 codones está indicando que esas cadenas de tres bases no son un precursor de un aminoácido, sino una indicación de terminación de la cadena polipeptídica (luego ampliaremos sobre esto). A la derecha se puede ver el detalle químico de las bases pirimídicas (timina, citosina y uracilo), una de las dos tipos de bases nitrogenadas que componen en ADN.
En la figura inferior tenemos, en la parte de arriba, un detalle químico de las bases purínicas (adenina, guanina), las otras bases nitrogenadas que componen en ADN. En la parte de abajo muestra una tabla están encadenados los componentes que forman los nucleósidos (base nitrogenada + pentosa (azúcar)), y por otro lado, la formación de los nucleótidos (nucleósido + ácido fosfórico), que son los monómeros de los ácidos nucleicos (ADN y ARN), y son utilizados para proveer la gran energía que tienen acumulada en sus enlaces de fosfato. Las células poseen enzimas (proteínas especializadas) cuya función es, precisamente, romper estos enlaces y proveer la energía necesaria para el metabolismo celular.
En la figura anterior podemos apreciar una cadena de ADN (con un detalle de su química a la izquierda, donde se ven sus componentes: la base nitrogenada, el azúcar de cinco átomos de carbono, y el grupo fosfato). Las cadenas que componen el ADN (ilustración de la derecha) son complementarias y antiparalelas (una va en la dirección 5' → 3' y la otra 3' → 5')
La actividad del ADN es modulada por proteínas que se unen a él en lugares precisos. Su molécula no solo guarda información genética; también posee, como parte de su estructura, señales para el control de su propia actividad.
El ADN nuclear de las células eucariotas (con núcleo) está en los cromosomas, cada uno de los cuales, aloja una molécula de ADN (dos cuando se replica). Estas grandes moléculas están empaquetadas en complejos núcleo-proteínicos: la cromatina → profase → cromosomas (formaciones discretas con máxima densidad en la metafase).
El complejo núcleo-proteico tiene: ADN - Histonas - Polipéptidos con funciones estructurales - Proteínas regulatorias de la actividad genética y las Enzimas para la síntesis y procesamiento de los ácidos nucleicos. (figura)
En la cromatina, el ADN, a intervalos regulares da dos vueltas sobre el núcleo constituido por un octómero de histonas (las dos vueltas abarcan 146 pares de bases nitrogenadas)
Histonas + superhélice → Nucleosoma
Los Nucleosomas están conectados por un tramo de ADN de unas 50 bases de longitud. Estas estructuras se encuentran extendidas durante la internase; al iniciarse la mitosis (proceso de duplicación) se empaquetan en un 'solenoide' de 6 nucleosomas por vuelta.
El patrimonio genético o genoma de cada individuo está contenido en las moléculas de ADN de sus cromosomas y Mitocondrias (10%). En los cromosomas están las unidades de información: los genes, que son porciones de ADN que dirigen la síntesis de distintas especies de ARN funcionantes: Ribosomal (ARNr), de Transferencia (ARNt), Mensajero (ARNm) y otros.
Replicación ADN ⇾ transcripción ⇾ ARNm ⇾ traducción ⇾ Proteína
El ciclo celular (división) es regulado por un complejo de proteínas, ciclinas y quinasas dependientes de ciclinas, activadas por agentes externos (factores de crecimiento) que se unen a receptores específicos en la membrana plasmática y transmiten señales al interior de la célula. Fases: G1, S, G2, M. La replicación del ADN ocurre durante la fase S.
Replicación: en primer lugar se separan las dos cadenas, comenzando en un punto fijo: sitio de origen, en el cual, secuencias específicas sirven de señales de reconocimiento para las enzimas y factores que inician la replicación. En las eucariotas comienza la replicación en varios sitios. La separación se inicia simultáneamente en todos los cromosomas y en muchos puntos en cada uno de ellos. Así se producen, en la doble hélice, botones o 'burbujas' (replicones). En el humano se forman alrededor de 50.000 burbujas.
El código genético tiene validez universal, o sea, los codones (los tripletes de bases de la tabla) tienen el mismo significado en todos los seres vivos. Los genes que codifican para proteínas contienen información que, transcrita al ARNm, sirve para dirigir el ordenamiento de aminoácidos de los polipéptidos correspondientes. El conjunto de codones con la información completa para la síntesis de una cadena polipeptídica, se denomina: cistrón.
En los animales superiores hay un gran exceso de ADN, en relación a la necesidad de codificación, ya que el número de genes que dirige la producción de ARNm en el ser humano, es menor de 100.000. A diferencia de las bacterias, las células eucariotas no tienen la información genética dispuesta en trazos continuos de ADN, sino dividida en segmentos denominados: exones. Entre ellos están los intrones (sin información). Los intrones son más que los exones. (figura)
ADN Mitocondrial: las mitocondrias tienen su propio genoma, con un total de 16.569 pares de bases en el humano. Tiene 37 genes; 2 codifican el ARN ribosomal, 22 en ARN transferencia, y los otros 13, subunidades polipeptídicas componentes del complejo de la cadena respiratoria. Este genoma no es autosuficiente. La mayoría de las proteínas mitocondriales son codificadas en el núcleo y luego transportadas a la organela.
Diferencias con el ADN nuclear: 1) no posee intrones; 2) no procede en partes iguales de ambos padres, solo de la madre; 3) se divide independientemente del ADN nuclear; 4) su código difiere del universal (figura); y 5) tiene mayor tasa de mutaciones (10 a 100 veces más). No tiene buen sistema de reparación.
La figura muestra las diferencias entre los códigos del ADN nuclear y mitocondrial. Referencias: U = uracilo, G = guanina, A= adenina. Como vemos se dan distintas variantes: lo que en el nuclear es una terminación, en el mitocondrial es un aminoácido; lo que el nuclear es un aminoácido, en el mitocondrial es una terminación; finalmente, el mismo codón significa distinto aminoácido en un que en otro.
ARNm: Se sintetiza durante la transcripción y la secuencia de sus nucleótidos es complementaria con una del las 'hebras' del ADN guía (nuclear). Como el ARNm se sintetiza sobre una de las cadenas del ADN, se dice que la transcripción es 'asimétrica'. La hebra 'molde' de ADN sobre la cual se ensambla el ARN complementario es llamada: 'antisentido' o no codificante. La otra, no transcrita tiene la misma secuencia que el ARN sintetizado (admitiendo el cambio de timina (T) por uracilo (U)) y es la hebra 'con sentido' o codificante.
En las eucariotas se transcribe cada gen en toda su extensión. Este ARN es el ARNm 'precursor'. El proceso post-transcripción del ARNm no termina con el agregado del 'capuchón' de 7-metilGTP en el extremo 5' y de la cola de poliA (adenosina) en el 3'. (ver figura) La gran mayoría del ARNm primario contiene secuencias intercaladas o intrones, que es necesario eliminar. Este proceso se llama 'splicing'. Se seccionan los intrones y luego se empalma en el orden correcto, los exornes para formar el ARNm 'maduro'. Los intrones tienen una extensión de 60 a 10.000 pares. Todos empiezan con las bases GU {1000 en binario, según la siguiente convención: C = 01, G = 10, A = 11, T = U = 00} (extremo 5'), y terminan en AG {1110} (extremo 3'). Hay otra secuencia 'condenso' situadas entre -20 y -50 del extremo 3' (aguas arriba) del intrón llamado 'ramificación' (UACUAAC en las levaduras).
Una vez formado el ARN maduro o mensajero, es exportado al citoplasma a través de los poros de la membrana nuclear.
La molécula de ARNm está formada por el 'capuchon' de 7-metilGTP en el extremo 5', ⨁ una secuencia no codifican (de más de 100 bases) hasta llegar al codón de iniciación, que siempre es AUG (metionina) ⨁ la porción codificante (sucesión de codones que indica la secuencia de aminoácidos en la proteína a sintetizar) ⨁ codón de terminación (UAA, UAG o UGA) ⨁ segmento de longitud variable ('trailer') ⨁ una 'cola' PoliA de 200 unidades de Adenina (en la mayoría de los ARNm). Las últimas dos porciones no son traducibles. El trozo codifican se denomina: 'encuadre' o 'marco de lectura abierto.
ARNr (ribosomal): integra los ribosomas. Las células de los mamíferos tienen una 1010 ribosomas; las bacterias 20.000. El ARNr maduro se une a las proteínas para constituir las subunidades de los ribosomas. Cuando las dos porciones se unen para formar la partícula ribosomal, queda entre ellas una hendidura por donde se desliza ('lector') el ARNm durante la síntesis de proteínas. En la partícula completa se reconocen dos sitios adyacentes. A (Aminoacil) y P (Peptidil) a las que se unen las moléculas de ARNt cargadas con aminoácido.
ARNt (transferencia): llamado soluble es el encargado de unirse a aminoácidos (aa) libres en el citosol y transportarlos hasta el lugar de us ensamble. Existen ARNt específicos para cada aa. Su extremo 3' se une al aa y es idéntico en todos los ARNt; la secuencia terminal es siempre CCA. La especificidad del ARNt radica en su anticodón; triplete complementario del codón del aa transportado. El apareamiento codón-anticodón [la LT propone una tabla y un método para encontrar el anticodón de cualquier codón a traves de seis ejes de simetría] es antiparalelo y la secuencia se lee en el sentido 5'→ 3'. Así GUA (anticodón)↔︎UAC (codón Tirosina), GGC (anticodón)↔︎GCC (codón Alanina).
El ARNt actua como una molécula intermediaria o adaptadora que reconoce una secuencia determinada de nucleótidos en el ARNm (codón) y permite ubicar en su sitio el aa correspondiente. ☛El ARN puede almacenar información genética y además catolizar su propia replicación.☚
Biosíntesis de las proteínas (traducción):
① Activación de aminoácidos: requiere aa libres, ARNt sintetasas, ARNt, ATP y Mg++.
El aa reacciona co el ATP → aminoacilAMP-enzima. El aa activado es transferido al ARNt específico.
② Iniciación: varios factores de iniciación interaccionan con el capuchón del extremo 5' del ARNm. También se unen subunidades ribosomales menores y otros, y forman el 'complejo de preiniciación'. Este complejo se desplaza sobre el ARNm en sentido 5' → 3' hasta ubicar el codón de iniciación AUG (Metionina), el cual se aparea al anticodón (CAU) del ARNti, junto a otros agregados, se adhiere al sitio P. Se forma el complejo de iniciación.
③ Elongación: es un ciclo que se repite con cada aa agregado. Se une al sitio A del ARNt, cuyo anticodón se acople al codón del ARNm → formación unión peptídica y translocación: el grupo carboxilo de la Metionina del ARNti; en el sitio P se une a la función amina del aa del ARNt en A. Se forma un dipeptidil que queda unido al ARNt ubicado en A. El ARNti del sitio P descargado de su Metionina es liberado. El dipeptidil se desplaza de A → P. El ribosoma avanza al siguiente codón sobre el ARNm en el sentido 5' → 3' y se repite el ciclo. Pasados varios ciclos, la cadena peptídica unida al ARNt en el sitio P ha crecido. La proteína se ensambla desde el extremo N al C-teminal. La cadena es transferida al ARNt en A.
④ Terminación: una vez completada la cadena se llaga a un codón de terminación (UAA, UAG o UGA) desconocido por el factor de liberación asociado a GTP. La cadena polipeptídica es separada del ARNt por hidrólisis. Se liberan el ARNt descargado y las subunidades ribosómicas. Una cadena de ARNm dirige simultáneamente la síntesis de varias moléculas de proteína. El conjunto de ocho o más ribosomas sobre el ARNm se llama 'polisoma'. La cadena debe plegarse sobre sí misma para adquirir una estructura adecuada (funcional). Este plegamiento es asistido por las 'chaperonas'.
Mutaciones genéticas: mutación es cualquier cambio en la secuencia del ADN.
a) Mutaciones puntuales: reemplazo de una base por otra, o la adición o pérdida de un par de bases. Sustitución: se cambia una base por otra del mismo tipo → Transcripción; se cambia una base por otra de otro tipo → Transversión. La sustitución de bases o la pérdida o adición puede perderse el marco de lectura abierta.
b) Cambios estructurales en los cromosomas: alteran una porción más o menos amplia del cromosoma y alteran el orden lineal del ADN.
1) Deleción: pérdida de un segmento de ADN.
2) Duplicación: repetición de un trozo de ADN.
3) Inversión: cambio de sentido de un tramo dentro de la molécula de ADN.
4) Traslocación: un segmento de una molécula de ADN cambia de posición relativa dentro del mismo cromosoma o se incorpora a otro.
Mutaciones somáticas y germinales: las mutaciones ocurren al azar en cualquier sitio del genoma y en cualquier célula del organismo. Las somáticas afectan al tejido que forman. Si la reproducción se hace por cruzamiento sexual la mutación desaparece con la muerte del organismo. Las mutaciones de las células germinales se transmiten a las nuevas generaciones; estas constituyen en última instancia, el origen de la variabilidad genética existente [esto es un prejuicio del neodarwinismo] en las poblaciones. Las mutaciones dan origen a formas alternativas del mismo gen, denominados 'a lelos'. En los organismos diploides {con doble número de cromosomas, a diferencia de los gametos que tienen una sola serie} los descendientes reciben, para los genes que ocupan el mismo locus {lugar} en un par de cromosomas homólogos, un alelo materno y otro paterno. Si uno es normal y el otro mutante: 'heterocigota' para ese caracter, si ambos son normales o ambos tienen igual mutación: 'homocigota'.
¡Nos vemos mañana!